南海所对南海抗生素抗性基因ARGs的多样性和空间分布特征研究取得重要进展
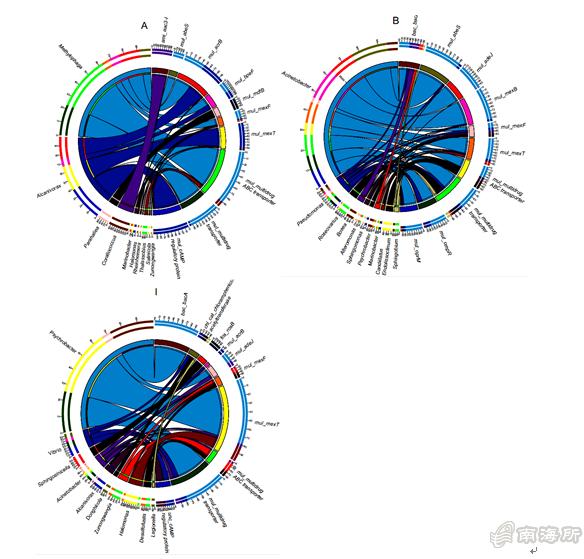
图1 优势ARGs及其贡献菌群
日前,中国水产科学研究院南海水产研究所南海渔业微生物资源挖掘与利用课题组开展南海近海海域、深远海和主要岛礁泻湖环境中抗生素抗性基因(ARGs)的多样性和空间分布特征的研究取得重要进展,为进一步加强和推进南海海洋微生物资源的挖掘利用及其种源潜在风险评估提供了重要参考。相关研究成果以题为“Diversity, abundances and distribution of antibiotic resistance genes and virulence factors in the South China Sea revealed by metagenomic sequencing”发表于环境领域国际知名期刊《Science of the Total Environment》(中科院1区、IF 10.753,苏浩昌为第一作者、曹煜成为通讯作者)。
该研究揭示了我国南海近海海域、深远海和主要岛礁泻湖环境中ARGs的全谱信息及其丰度等空间分布特征,共检测出16类140种ARGs,其中多重耐药ARGs丰度最高,为120.8 ppm,其次为杆菌肽类、氨基糖苷类、beta内酰胺类、大环内酯类、四环素、氯霉素类和磺胺类ARGs。Circos分析结果显示,对近海海域、深远海及岛礁泻湖区中ARGs的最大贡献者分别为Methylophaga、Acinetobacter、Psychrobacter,它们的贡献率分别为41%、56%、25%;就ARGs总丰度而言,近海海域为312.9 ppm,比深远海和岛礁区高1.86-2.39倍(p < 0.05)。RDA分析结果显示,硝酸盐、铅等环境因子与南海ARGs的丰度呈显著正相关关系(p < 0.05)。NMDS分析结果显示,南海海域ARGs的多样性和丰度与地理位置显著相关,以近海、深远海和岛礁区进行聚类。上述研究结果表明,人类活动可能对南海ARGs的丰度和空间分布产生重要影响,因而在南海深远海及岛礁区进行海洋渔业微生物资源的挖掘利用,在一定程度上有利于规避ARGs潜在风险干扰。
该研究得到中国水产科学研究院科技创新团队项目(2020TD54)、广东省基础与应用基础研究项目(2019B030302004)、农业财政专项“南海生物资源调查与评估”(NFZX2021)等项目资助。
论文链接:https://doi.org/10.1016/j.scitotenv.2021.152803
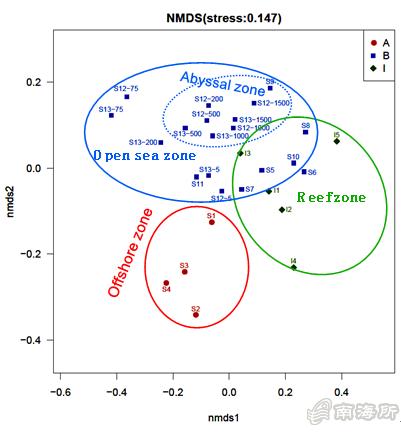
图2 ARGs丰度的NMDS分析
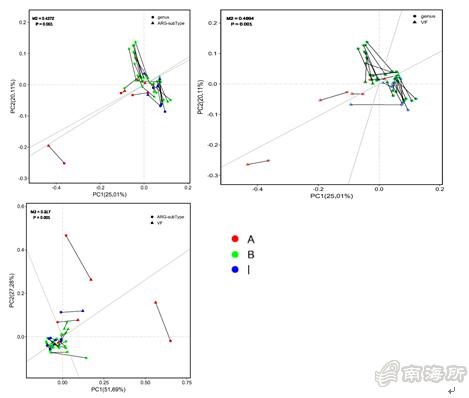
图3 ARGs丰度与菌群的Procrustes分析
分享到朋友圈




 粤公网安备44010502001742号
粤公网安备44010502001742号